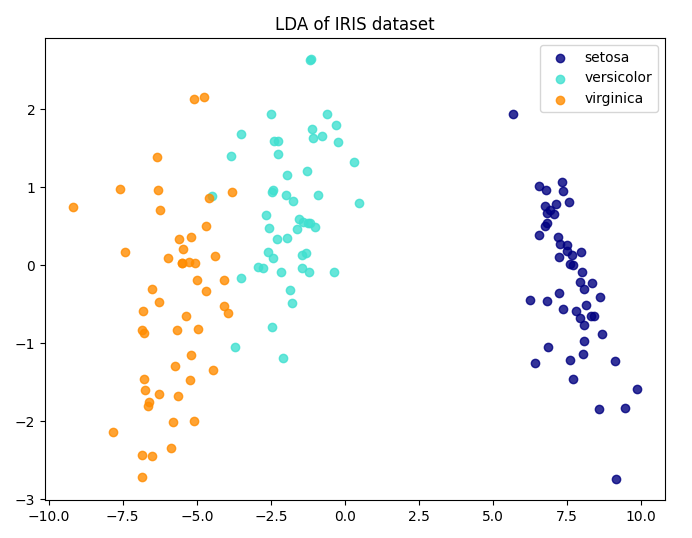
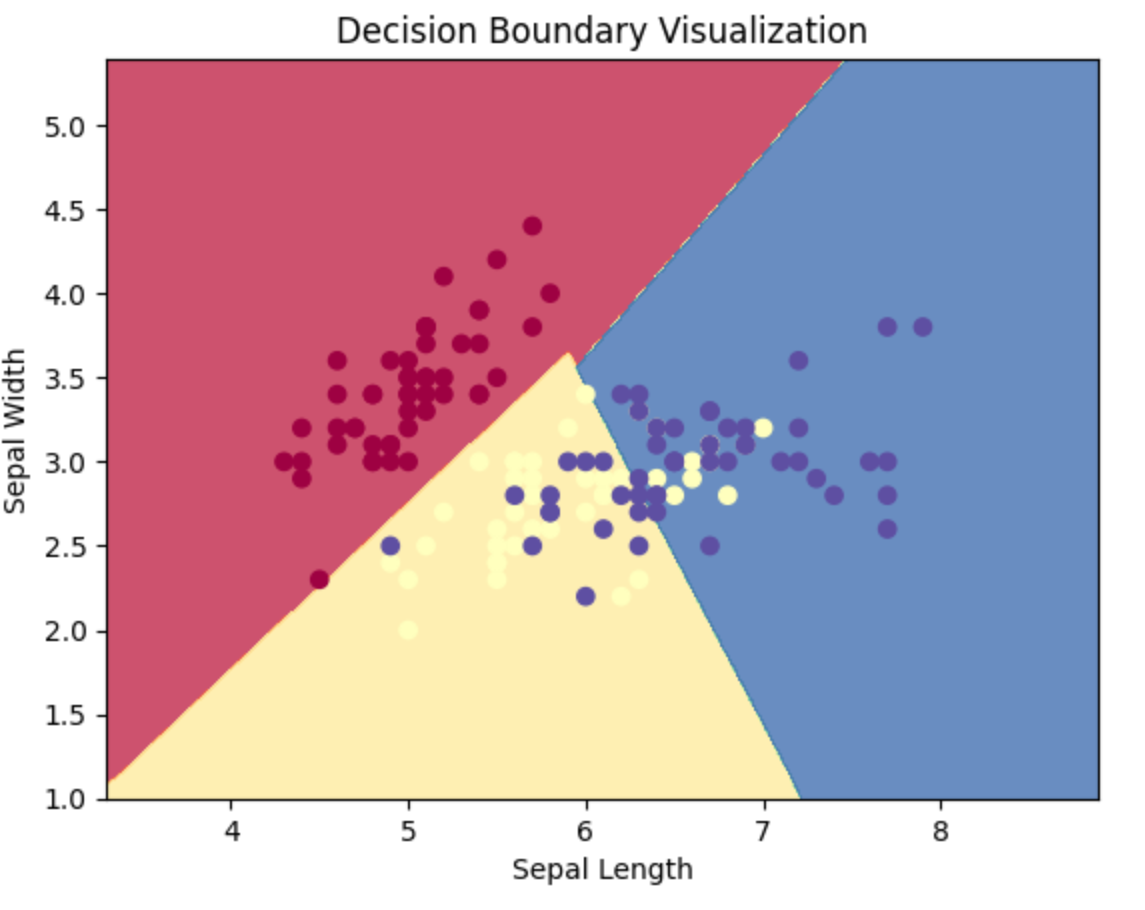
python机器学习-模型评估与参数调优
性能指标(二分类问题)
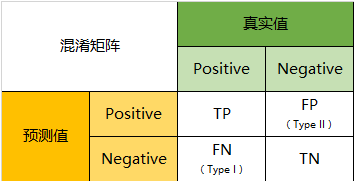
混淆矩阵
首先,为什么在有准确率(accuracy)的情况下,还要引入别的指标呢?因为受困于数据收集的客观限制,容易出现不平衡问题,比如正类的数量远远大于负类。这样,即使模型将所有样本预测为正类,损失函数也很低,达到了欺骗的效果。因此,我们需要将正类和负类分别的预测结果列出来,如混淆矩阵。
准确率和召回率
基于混淆矩阵,得到了准确率(precision)和召回率(recall):
precision = TP/(TP+FP)
recall = TP/(TP+FN)
ROC 曲线
我们通过真正率(TPR)和假真率(FPR)来衡量分类器的性能。
通过 ROC 空间,我们明白了一条 ROC 曲线其实代表了无数个分类器。那么我们为什么常常用一条 ROC 曲线来描述一个分类器呢?仔细观察 ROC 曲线,发现其都是上升的曲线(斜率大于 0),且都通过点(0,0)和点(1,1)。其实,这些点代表着一个分类器在不同阈值下的分类效果,具体的,曲线从左往右可以认为是阈值从 0 到 1 的变化过程。当分类器阈值为 0,代表不加以识别全部判断为 0,此时 TP=FP=0,TPR=TP/P=0,FPR=FP/N=0;当分类器阈值为 1,代表不加以识别全部判断为 1,此时 FN=TN=0,P=TP+FN=TP, TPR=TP/P=1,N=FP+TN=FP, FPR=FP/N=1。所以,ROC 曲线描述的其实是分类器性能随着分类器阈值的变化而变化的过程。对于 ROC 曲线,一个重要的特征是它的面积,面积为 0.5 为随机分类,识别能力为 0,面积越接近于 1 识别能力越强,面积等于 1 为完全识别。